パフォーマンスの向上#
このチュートリアルのこの部分では、Cython、Numba、およびpandas.eval()を使用して、pandas DataFrameで操作する特定の関数の速度を上げる方法を調査します。一般に、CythonとNumbaを使用すると、pandas.eval()を使用するよりも大きな速度向上が期待できますが、はるかに多くのコードが必要になります。
注
このチュートリアルの手順に従うことに加えて、パフォーマンスの向上に関心のあるユーザーは、pandasの推奨される依存関係をインストールすることを強くお勧めします。これらの依存関係は通常デフォルトではインストールされませんが、存在すれば速度向上が期待できます。
Cython (pandas 用 C 拡張機能の作成)#
多くのユースケースでは、純粋な Python と NumPy で pandas を記述するだけで十分です。しかし、計算量の多いアプリケーションでは、Cython に作業をオフロードすることで、かなりの速度向上を実現できる可能性があります。
このチュートリアルでは、for ループを削除し、NumPy のベクトル化を利用するなど、Python で可能な限りリファクタリングを終えていることを前提としています。最初に Python で最適化を行うことは常に価値があります。
このチュートリアルでは、遅い計算を Cython 化する「典型的な」プロセスを説明します。Cython ドキュメントの例を pandas のコンテキストで使用します。最終的な Cython 化されたソリューションは、純粋な Python ソリューションよりも約 100 倍高速です。
純粋なPython#
関数を行ごとに適用したい DataFrame があります。
In [1]: df = pd.DataFrame(
...: {
...: "a": np.random.randn(1000),
...: "b": np.random.randn(1000),
...: "N": np.random.randint(100, 1000, (1000)),
...: "x": "x",
...: }
...: )
...:
In [2]: df
Out[2]:
a b N x
0 0.469112 -0.218470 585 x
1 -0.282863 -0.061645 841 x
2 -1.509059 -0.723780 251 x
3 -1.135632 0.551225 972 x
4 1.212112 -0.497767 181 x
.. ... ... ... ..
995 -1.512743 0.874737 374 x
996 0.933753 1.120790 246 x
997 -0.308013 0.198768 157 x
998 -0.079915 1.757555 977 x
999 -1.010589 -1.115680 770 x
[1000 rows x 4 columns]
純粋なPythonの関数は次のとおりです。
In [3]: def f(x):
...: return x * (x - 1)
...:
In [4]: def integrate_f(a, b, N):
...: s = 0
...: dx = (b - a) / N
...: for i in range(N):
...: s += f(a + i * dx)
...: return s * dx
...:
DataFrame.apply()(行ごと)を使用して結果を得ます。
In [5]: %timeit df.apply(lambda x: integrate_f(x["a"], x["b"], x["N"]), axis=1)
80.3 ms +- 1.18 ms per loop (mean +- std. dev. of 7 runs, 10 loops each)
prun ipython magic 関数を使って、この操作中にどこで時間が費やされているかを見てみましょう。
# most time consuming 4 calls
In [6]: %prun -l 4 df.apply(lambda x: integrate_f(x["a"], x["b"], x["N"]), axis=1) # noqa E999
605956 function calls (605938 primitive calls) in 0.173 seconds
Ordered by: internal time
List reduced from 163 to 4 due to restriction <4>
ncalls tottime percall cumtime percall filename:lineno(function)
1000 0.101 0.000 0.154 0.000 <ipython-input-4-c2a74e076cf0>:1(integrate_f)
552423 0.053 0.000 0.053 0.000 <ipython-input-3-c138bdd570e3>:1(f)
3000 0.003 0.000 0.013 0.000 series.py:1104(__getitem__)
3000 0.002 0.000 0.006 0.000 series.py:1229(_get_value)
時間の大部分は圧倒的に integrate_f または f 内で費やされているため、これら 2 つの関数の Cython 化に集中します。
プレーンなCython#
まず、Cython マジック関数を IPython にインポートする必要があります。
In [7]: %load_ext Cython
次に、関数をCythonにコピーしてみましょう。
In [8]: %%cython
...: def f_plain(x):
...: return x * (x - 1)
...: def integrate_f_plain(a, b, N):
...: s = 0
...: dx = (b - a) / N
...: for i in range(N):
...: s += f_plain(a + i * dx)
...: return s * dx
...:
In [9]: %timeit df.apply(lambda x: integrate_f_plain(x["a"], x["b"], x["N"]), axis=1)
48.7 ms +- 490 us per loop (mean +- std. dev. of 7 runs, 10 loops each)
これにより、純粋な Python アプローチと比較してパフォーマンスが 3 分の 1 向上しました。
C型の宣言#
関数の変数と戻り値の型に注釈を付けたり、cdef と cpdef を使用してパフォーマンスを向上させることができます。
In [10]: %%cython
....: cdef double f_typed(double x) except? -2:
....: return x * (x - 1)
....: cpdef double integrate_f_typed(double a, double b, int N):
....: cdef int i
....: cdef double s, dx
....: s = 0
....: dx = (b - a) / N
....: for i in range(N):
....: s += f_typed(a + i * dx)
....: return s * dx
....:
In [11]: %timeit df.apply(lambda x: integrate_f_typed(x["a"], x["b"], x["N"]), axis=1)
7.5 ms +- 29.6 us per loop (mean +- std. dev. of 7 runs, 100 loops each)
C型で関数に注釈を付けると、元のPython実装と比較してパフォーマンスが10倍以上向上します。
ndarray の使用#
再プロファイリングすると、各行から Series を作成し、インデックスとシリーズの両方から __getitem__ を呼び出す(各行につき 3 回)のに時間が費やされます。これらの Python 関数呼び出しはコストが高く、np.ndarray を渡すことで改善できます。
In [12]: %prun -l 4 df.apply(lambda x: integrate_f_typed(x["a"], x["b"], x["N"]), axis=1)
52533 function calls (52515 primitive calls) in 0.019 seconds
Ordered by: internal time
List reduced from 161 to 4 due to restriction <4>
ncalls tottime percall cumtime percall filename:lineno(function)
3000 0.003 0.000 0.012 0.000 series.py:1104(__getitem__)
3000 0.002 0.000 0.005 0.000 series.py:1229(_get_value)
3000 0.002 0.000 0.003 0.000 indexing.py:2765(check_dict_or_set_indexers)
3000 0.002 0.000 0.002 0.000 base.py:3784(get_loc)
In [13]: %%cython
....: cimport numpy as np
....: import numpy as np
....: cdef double f_typed(double x) except? -2:
....: return x * (x - 1)
....: cpdef double integrate_f_typed(double a, double b, int N):
....: cdef int i
....: cdef double s, dx
....: s = 0
....: dx = (b - a) / N
....: for i in range(N):
....: s += f_typed(a + i * dx)
....: return s * dx
....: cpdef np.ndarray[double] apply_integrate_f(np.ndarray col_a, np.ndarray col_b,
....: np.ndarray col_N):
....: assert (col_a.dtype == np.float64
....: and col_b.dtype == np.float64 and col_N.dtype == np.dtype(int))
....: cdef Py_ssize_t i, n = len(col_N)
....: assert (len(col_a) == len(col_b) == n)
....: cdef np.ndarray[double] res = np.empty(n)
....: for i in range(len(col_a)):
....: res[i] = integrate_f_typed(col_a[i], col_b[i], col_N[i])
....: return res
....:
Content of stderr:
In file included from /home/runner/micromamba/envs/test/lib/python3.10/site-packages/numpy/core/include/numpy/ndarraytypes.h:1929,
from /home/runner/micromamba/envs/test/lib/python3.10/site-packages/numpy/core/include/numpy/ndarrayobject.h:12,
from /home/runner/micromamba/envs/test/lib/python3.10/site-packages/numpy/core/include/numpy/arrayobject.h:5,
from /home/runner/.cache/ipython/cython/_cython_magic_1f8c1b875aeb076a8ef75ac5199664d0fea77dfb626f30a4e36b3263c3db7ec2.c:1138:
/home/runner/micromamba/envs/test/lib/python3.10/site-packages/numpy/core/include/numpy/npy_1_7_deprecated_api.h:17:2: warning: #warning "Using deprecated NumPy API, disable it with " "#define NPY_NO_DEPRECATED_API NPY_1_7_API_VERSION" [-Wcpp]
17 | #warning "Using deprecated NumPy API, disable it with " \
| ^~~~~~~
この実装では、ゼロの配列を作成し、各行に適用された integrate_f_typed の結果を挿入します。ndarray をループする方が、Cython では Series オブジェクトをループするよりも高速です。
apply_integrate_fはnp.ndarrayを受け入れるように型付けされているため、この関数を利用するにはSeries.to_numpy()の呼び出しが必要です。
In [14]: %timeit apply_integrate_f(df["a"].to_numpy(), df["b"].to_numpy(), df["N"].to_numpy())
830 us +- 945 ns per loop (mean +- std. dev. of 7 runs, 1,000 loops each)
パフォーマンスは以前の実装からほぼ10倍向上しました。
コンパイラディレクティブの無効化#
時間の大部分は現在 apply_integrate_f で費やされています。Cython の boundscheck と wraparound チェックを無効にすると、さらなるパフォーマンス向上が期待できます。
In [15]: %prun -l 4 apply_integrate_f(df["a"].to_numpy(), df["b"].to_numpy(), df["N"].to_numpy())
78 function calls in 0.001 seconds
Ordered by: internal time
List reduced from 21 to 4 due to restriction <4>
ncalls tottime percall cumtime percall filename:lineno(function)
1 0.001 0.001 0.001 0.001 <string>:1(<module>)
1 0.000 0.000 0.001 0.001 {built-in method builtins.exec}
3 0.000 0.000 0.000 0.000 frame.py:4067(__getitem__)
3 0.000 0.000 0.000 0.000 base.py:545(to_numpy)
In [16]: %%cython
....: cimport cython
....: cimport numpy as np
....: import numpy as np
....: cdef np.float64_t f_typed(np.float64_t x) except? -2:
....: return x * (x - 1)
....: cpdef np.float64_t integrate_f_typed(np.float64_t a, np.float64_t b, np.int64_t N):
....: cdef np.int64_t i
....: cdef np.float64_t s = 0.0, dx
....: dx = (b - a) / N
....: for i in range(N):
....: s += f_typed(a + i * dx)
....: return s * dx
....: @cython.boundscheck(False)
....: @cython.wraparound(False)
....: cpdef np.ndarray[np.float64_t] apply_integrate_f_wrap(
....: np.ndarray[np.float64_t] col_a,
....: np.ndarray[np.float64_t] col_b,
....: np.ndarray[np.int64_t] col_N
....: ):
....: cdef np.int64_t i, n = len(col_N)
....: assert len(col_a) == len(col_b) == n
....: cdef np.ndarray[np.float64_t] res = np.empty(n, dtype=np.float64)
....: for i in range(n):
....: res[i] = integrate_f_typed(col_a[i], col_b[i], col_N[i])
....: return res
....:
Content of stderr:
In file included from /home/runner/micromamba/envs/test/lib/python3.10/site-packages/numpy/core/include/numpy/ndarraytypes.h:1929,
from /home/runner/micromamba/envs/test/lib/python3.10/site-packages/numpy/core/include/numpy/ndarrayobject.h:12,
from /home/runner/micromamba/envs/test/lib/python3.10/site-packages/numpy/core/include/numpy/arrayobject.h:5,
from /home/runner/.cache/ipython/cython/_cython_magic_344a9e4468707236d239faf5bdfacf0d14a35efa7e89d2a7b09ae36b339492db.c:1139:
/home/runner/micromamba/envs/test/lib/python3.10/site-packages/numpy/core/include/numpy/npy_1_7_deprecated_api.h:17:2: warning: #warning "Using deprecated NumPy API, disable it with " "#define NPY_NO_DEPRECATED_API NPY_1_7_API_VERSION" [-Wcpp]
17 | #warning "Using deprecated NumPy API, disable it with " \
| ^~~~~~~
In [17]: %timeit apply_integrate_f_wrap(df["a"].to_numpy(), df["b"].to_numpy(), df["N"].to_numpy())
624 us +- 2.43 us per loop (mean +- std. dev. of 7 runs, 1,000 loops each)
ただし、ループインデクサ i が配列内の無効な位置にアクセスすると、メモリアクセスがチェックされないため、セグメンテーションフォールトが発生します。boundscheck と wraparound の詳細については、Cython のコンパイラディレクティブに関するドキュメントを参照してください。
Numba (JIT コンパイル)#
Cython コードを静的にコンパイルする代わりに、Numba を使用して動的なジャストインタイム (JIT) コンパイラを使用する方法があります。
Numbaを使用すると、@jitで関数をデコレートすることで、C、C++、Fortranと同様のパフォーマンスでネイティブマシン命令にJITコンパイルできる純粋なPython関数を記述できます。
Numba は、インポート時、実行時、または静的 (付属の pycc ツールを使用) に LLVM コンパイラインフラストラクチャを使用して最適化されたマシンコードを生成することで機能します。Numba は、Python を CPU または GPU ハードウェアで実行するためのコンパイルをサポートしており、Python の科学ソフトウェアスタックと統合するように設計されています。
注
@jit コンパイルは関数の実行時にオーバーヘッドを追加するため、特に小さなデータセットを使用する場合はパフォーマンスのメリットが実現されない可能性があります。関数が実行されるたびにコンパイルのオーバーヘッドを避けるために、関数のキャッシュを検討してください。
Numba は pandas と 2 つの方法で使用できます。
一部のpandasメソッドで
engine="numba"キーワードを指定する@jitでデコレートされた独自の Python 関数を定義し、SeriesまたはDataFrameの基盤となる NumPy 配列 (Series.to_numpy()を使用) を関数に渡します。
pandas Numba エンジン#
Numba がインストールされている場合、一部の pandas メソッドで engine="numba" を指定することで、Numba を使用してメソッドを実行できます。engine="numba" をサポートするメソッドには、engine_kwargs キーワードもあり、これには辞書を受け入れ、@jit デコレータに渡すブール値を持つ "nogil"、"nopython"、"parallel" キーを指定できます。engine_kwargs が指定されていない場合、特に指定がない限り、デフォルトは {"nogil": False, "nopython": True, "parallel": False} です。
注
パフォーマンスに関して、Numba エンジンを使用して関数を初めて実行する場合、Numba の関数コンパイルオーバーヘッドがあるため、処理が遅くなります。しかし、JIT コンパイルされた関数はキャッシュされるため、その後の呼び出しは高速になります。一般的に、Numba エンジンは大量のデータポイント(例えば 100 万以上)で高性能を発揮します。
In [1]: data = pd.Series(range(1_000_000)) # noqa: E225
In [2]: roll = data.rolling(10)
In [3]: def f(x):
...: return np.sum(x) + 5
# Run the first time, compilation time will affect performance
In [4]: %timeit -r 1 -n 1 roll.apply(f, engine='numba', raw=True)
1.23 s ± 0 ns per loop (mean ± std. dev. of 1 run, 1 loop each)
# Function is cached and performance will improve
In [5]: %timeit roll.apply(f, engine='numba', raw=True)
188 ms ± 1.93 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
In [6]: %timeit roll.apply(f, engine='cython', raw=True)
3.92 s ± 59 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
計算ハードウェアに複数の CPU が搭載されている場合、parallel を True に設定して 1 つ以上の CPU を活用することで、最大のパフォーマンス向上を実現できます。内部的に、pandas は numba を利用して DataFrame の列に対して計算を並列化します。したがって、このパフォーマンス向上は、多数の列を持つ DataFrame の場合にのみ有益です。
In [1]: import numba
In [2]: numba.set_num_threads(1)
In [3]: df = pd.DataFrame(np.random.randn(10_000, 100))
In [4]: roll = df.rolling(100)
In [5]: %timeit roll.mean(engine="numba", engine_kwargs={"parallel": True})
347 ms ± 26 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
In [6]: numba.set_num_threads(2)
In [7]: %timeit roll.mean(engine="numba", engine_kwargs={"parallel": True})
201 ms ± 2.97 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
カスタム関数の例#
@jit でデコレートされたカスタムの Python 関数は、NumPy 配列表現を Series.to_numpy() で渡すことで pandas オブジェクトで使用できます。
import numba
@numba.jit
def f_plain(x):
return x * (x - 1)
@numba.jit
def integrate_f_numba(a, b, N):
s = 0
dx = (b - a) / N
for i in range(N):
s += f_plain(a + i * dx)
return s * dx
@numba.jit
def apply_integrate_f_numba(col_a, col_b, col_N):
n = len(col_N)
result = np.empty(n, dtype="float64")
assert len(col_a) == len(col_b) == n
for i in range(n):
result[i] = integrate_f_numba(col_a[i], col_b[i], col_N[i])
return result
def compute_numba(df):
result = apply_integrate_f_numba(
df["a"].to_numpy(), df["b"].to_numpy(), df["N"].to_numpy()
)
return pd.Series(result, index=df.index, name="result")
In [4]: %timeit compute_numba(df)
1000 loops, best of 3: 798 us per loop
この例では、Numba の方が Cython よりも高速でした。
Numba を使用すると、ユーザーがベクトルの観測値を明示的にループする必要のないベクトル化された関数も作成できます。ベクトル化された関数は各行に自動的に適用されます。各観測値を 2 倍にする次の例を考えてみましょう。
import numba
def double_every_value_nonumba(x):
return x * 2
@numba.vectorize
def double_every_value_withnumba(x): # noqa E501
return x * 2
# Custom function without numba
In [5]: %timeit df["col1_doubled"] = df["a"].apply(double_every_value_nonumba) # noqa E501
1000 loops, best of 3: 797 us per loop
# Standard implementation (faster than a custom function)
In [6]: %timeit df["col1_doubled"] = df["a"] * 2
1000 loops, best of 3: 233 us per loop
# Custom function with numba
In [7]: %timeit df["col1_doubled"] = double_every_value_withnumba(df["a"].to_numpy())
1000 loops, best of 3: 145 us per loop
注意点#
Numbaは、NumPy配列に数値関数を適用する関数の高速化に最も優れています。サポートされていないPythonまたはNumPyコードを含む関数を@jitしようとすると、コンパイルはオブジェクトモードに戻り、関数が高速化されない可能性が高くなります。コードを高速化する方法で関数をコンパイルできない場合にNumbaにエラーをスローさせたい場合は、Numbaに引数nopython=Trueを渡します(例:@jit(nopython=True))。Numbaモードのトラブルシューティングの詳細については、Numbaトラブルシューティングページを参照してください。
parallel=True (例: @jit(parallel=True)) を使用すると、スレッド層が安全でない動作につながる場合、SIGABRT が発生する可能性があります。parallel=True で JIT 関数を実行する前に、まず安全なスレッド層を指定してください。
一般的に、Numba の使用中にセグメンテーションフォールト (SIGSEGV) が発生した場合は、Numba の issue tracker に問題を報告してください。
eval() による式評価#
トップレベル関数 pandas.eval() は、Series および DataFrame の高性能な式評価を実装しています。式評価により、操作を文字列として表現でき、大規模な DataFrame の算術式と論理式を一度に評価することで、パフォーマンスが向上する可能性があります。
注
単純な式や小さなDataFrameを含む式にはeval()を使用すべきではありません。実際、eval()は、小さな式やオブジェクトに対しては、プレーンなPythonよりも何桁も遅くなります。良い経験則として、10,000行を超えるDataFrameがある場合にのみeval()を使用することをお勧めします。
サポートされる構文#
これらの操作は pandas.eval() によってサポートされています。
左シフト(
<<)および右シフト(>>)演算子を除く算術演算子。例:df + 2 * pi / s ** 4 % 42 - the_golden_ratio連鎖比較を含む比較演算子。例:
2 < df < df2ブール演算、例:
df < df2 and df3 < df4 or not df_boollistとtupleリテラル、例:[1, 2]または(1, 2)属性アクセス、例:
df.a添字式、例:
df[0]単純な変数評価、例:
pd.eval("df")(これはあまり役に立たない)数学関数:
sin,cos,exp,log,expm1,log1p,sqrt,sinh,cosh,tanh,arcsin,arccos,arctan,arccosh,arcsinh,arctanh,abs,arctan2およびlog10。
以下のPython構文は許可されません
式
数学関数以外の関数呼び出し。
is/is not演算if式lambda式list/set/dict内包表記リテラルな
dictおよびset式yield式ジェネレータ式
スカラー値のみで構成されるブール式
ステートメント
ローカル変数#
式で使用したいローカル変数は、その名前の前に @ 文字を付けて明示的に参照する必要があります。このメカニズムは、DataFrame.query() と DataFrame.eval() の両方で同じです。例:
In [18]: df = pd.DataFrame(np.random.randn(5, 2), columns=list("ab"))
In [19]: newcol = np.random.randn(len(df))
In [20]: df.eval("b + @newcol")
Out[20]:
0 -0.206122
1 -1.029587
2 0.519726
3 -2.052589
4 1.453210
dtype: float64
In [21]: df.query("b < @newcol")
Out[21]:
a b
1 0.160268 -0.848896
3 0.333758 -1.180355
4 0.572182 0.439895
ローカル変数の前に @ を付けないと、pandas は変数が未定義であるという例外を発生させます。
DataFrame.eval() と DataFrame.query() を使用する場合、これにより、式内で同じ名前のローカル変数と DataFrame 列を持つことができます。
In [22]: a = np.random.randn()
In [23]: df.query("@a < a")
Out[23]:
a b
0 0.473349 0.891236
1 0.160268 -0.848896
2 0.803311 1.662031
3 0.333758 -1.180355
4 0.572182 0.439895
In [24]: df.loc[a < df["a"]] # same as the previous expression
Out[24]:
a b
0 0.473349 0.891236
1 0.160268 -0.848896
2 0.803311 1.662031
3 0.333758 -1.180355
4 0.572182 0.439895
警告
そのコンテキストで @ プレフィックスを使用できない場合、pandas.eval() は例外を発生させます。
In [25]: a, b = 1, 2
In [26]: pd.eval("@a + b")
Traceback (most recent call last):
File ~/micromamba/envs/test/lib/python3.10/site-packages/IPython/core/interactiveshell.py:3579 in run_code
exec(code_obj, self.user_global_ns, self.user_ns)
Cell In[26], line 1
pd.eval("@a + b")
File ~/work/pandas/pandas/pandas/core/computation/eval.py:328 in eval
_check_for_locals(expr, level, parser)
File ~/work/pandas/pandas/pandas/core/computation/eval.py:170 in _check_for_locals
raise SyntaxError(msg)
File <string>
SyntaxError: The '@' prefix is not allowed in top-level eval calls.
please refer to your variables by name without the '@' prefix.
この場合、標準の Python と同じように変数に参照してください。
In [27]: pd.eval("a + b")
Out[27]: 3
pandas.eval() パーサー#
2つの異なる式構文パーサーがあります。
デフォルトの 'pandas' パーサーは、クエリのような操作(比較、論理積、論理和)を表現するためのより直感的な構文を可能にします。特に、& および | 演算子の優先順位は、対応する論理演算 and および or の優先順位と同じになります。
たとえば、上記の結合は括弧なしで記述できます。または、'python' パーサーを使用して厳密な Python セマンティクスを強制することもできます。
In [28]: nrows, ncols = 20000, 100
In [29]: df1, df2, df3, df4 = [pd.DataFrame(np.random.randn(nrows, ncols)) for _ in range(4)]
In [30]: expr = "(df1 > 0) & (df2 > 0) & (df3 > 0) & (df4 > 0)"
In [31]: x = pd.eval(expr, parser="python")
In [32]: expr_no_parens = "df1 > 0 & df2 > 0 & df3 > 0 & df4 > 0"
In [33]: y = pd.eval(expr_no_parens, parser="pandas")
In [34]: np.all(x == y)
Out[34]: True
同じ式は、 and という単語を使って「and」結合することもできます。
In [35]: expr = "(df1 > 0) & (df2 > 0) & (df3 > 0) & (df4 > 0)"
In [36]: x = pd.eval(expr, parser="python")
In [37]: expr_with_ands = "df1 > 0 and df2 > 0 and df3 > 0 and df4 > 0"
In [38]: y = pd.eval(expr_with_ands, parser="pandas")
In [39]: np.all(x == y)
Out[39]: True
pandas.eval() エンジン#
2つの異なる式エンジンがあります。
'numexpr' エンジンは、大規模な DataFrame の場合、標準の Python 構文と比較してパフォーマンスの向上をもたらすことができる、より高性能なエンジンです。このエンジンには、オプションの依存関係である numexpr がインストールされている必要があります。
'python' エンジンは、一般的に他の評価エンジンとのテストを除いては役立ちません。engine='python' でeval() を使用してもパフォーマンス上のメリットは一切なく、パフォーマンスが低下する可能性があります。
In [40]: %timeit df1 + df2 + df3 + df4
7.75 ms +- 186 us per loop (mean +- std. dev. of 7 runs, 100 loops each)
In [41]: %timeit pd.eval("df1 + df2 + df3 + df4", engine="python")
8.54 ms +- 73.2 us per loop (mean +- std. dev. of 7 runs, 100 loops each)
DataFrame.eval() メソッド#
トップレベルの pandas.eval() 関数に加えて、DataFrame の「コンテキスト」で式を評価することもできます。
In [42]: df = pd.DataFrame(np.random.randn(5, 2), columns=["a", "b"])
In [43]: df.eval("a + b")
Out[43]:
0 -0.161099
1 0.805452
2 0.747447
3 1.189042
4 -2.057490
dtype: float64
有効な pandas.eval() 式はすべて有効な DataFrame.eval() 式であり、評価対象の列に DataFrame の名前をプレフィックスとして付ける必要がないという利点があります。
さらに、式内で列の割り当てを実行できます。これにより、数式による評価が可能になります。割り当てのターゲットは新しい列名でも既存の列名でもよく、有効な Python 識別子である必要があります。
In [44]: df = pd.DataFrame(dict(a=range(5), b=range(5, 10)))
In [45]: df = df.eval("c = a + b")
In [46]: df = df.eval("d = a + b + c")
In [47]: df = df.eval("a = 1")
In [48]: df
Out[48]:
a b c d
0 1 5 5 10
1 1 6 7 14
2 1 7 9 18
3 1 8 11 22
4 1 9 13 26
新しい列または変更された列を持つDataFrameのコピーが返され、元のフレームは変更されません。
In [49]: df
Out[49]:
a b c d
0 1 5 5 10
1 1 6 7 14
2 1 7 9 18
3 1 8 11 22
4 1 9 13 26
In [50]: df.eval("e = a - c")
Out[50]:
a b c d e
0 1 5 5 10 -4
1 1 6 7 14 -6
2 1 7 9 18 -8
3 1 8 11 22 -10
4 1 9 13 26 -12
In [51]: df
Out[51]:
a b c d
0 1 5 5 10
1 1 6 7 14
2 1 7 9 18
3 1 8 11 22
4 1 9 13 26
複数行の文字列を使用することで、複数の列の割り当てを実行できます。
In [52]: df.eval(
....: """
....: c = a + b
....: d = a + b + c
....: a = 1""",
....: )
....:
Out[52]:
a b c d
0 1 5 6 12
1 1 6 7 14
2 1 7 8 16
3 1 8 9 18
4 1 9 10 20
標準のPythonでは同等なのは
In [53]: df = pd.DataFrame(dict(a=range(5), b=range(5, 10)))
In [54]: df["c"] = df["a"] + df["b"]
In [55]: df["d"] = df["a"] + df["b"] + df["c"]
In [56]: df["a"] = 1
In [57]: df
Out[57]:
a b c d
0 1 5 5 10
1 1 6 7 14
2 1 7 9 18
3 1 8 11 22
4 1 9 13 26
eval() パフォーマンス比較#
pandas.eval() は、大きな配列を含む式でうまく機能します。
In [58]: nrows, ncols = 20000, 100
In [59]: df1, df2, df3, df4 = [pd.DataFrame(np.random.randn(nrows, ncols)) for _ in range(4)]
DataFrame 算術
In [60]: %timeit df1 + df2 + df3 + df4
7.83 ms +- 224 us per loop (mean +- std. dev. of 7 runs, 100 loops each)
In [61]: %timeit pd.eval("df1 + df2 + df3 + df4")
3.16 ms +- 106 us per loop (mean +- std. dev. of 7 runs, 100 loops each)
DataFrame 比較
In [62]: %timeit (df1 > 0) & (df2 > 0) & (df3 > 0) & (df4 > 0)
5.53 ms +- 34 us per loop (mean +- std. dev. of 7 runs, 100 loops each)
In [63]: %timeit pd.eval("(df1 > 0) & (df2 > 0) & (df3 > 0) & (df4 > 0)")
8.68 ms +- 53.9 us per loop (mean +- std. dev. of 7 runs, 100 loops each)
軸が整列されていないDataFrameとの算術演算。
In [64]: s = pd.Series(np.random.randn(50))
In [65]: %timeit df1 + df2 + df3 + df4 + s
12.7 ms +- 83.8 us per loop (mean +- std. dev. of 7 runs, 100 loops each)
In [66]: %timeit pd.eval("df1 + df2 + df3 + df4 + s")
3.65 ms +- 45.4 us per loop (mean +- std. dev. of 7 runs, 100 loops each)
注
次のような操作
1 and 2 # would parse to 1 & 2, but should evaluate to 2
3 or 4 # would parse to 3 | 4, but should evaluate to 3
~1 # this is okay, but slower when using eval
Pythonで実行する必要があります。型がboolまたはnp.bool_ではないスカラーオペランドとのブール/ビット演算を実行しようとすると、例外が発生します。
計算に関わるフレームのサイズを関数とするpandas.eval()の実行時間を示すプロットです。2つの線は2つの異なるエンジンを表しています。
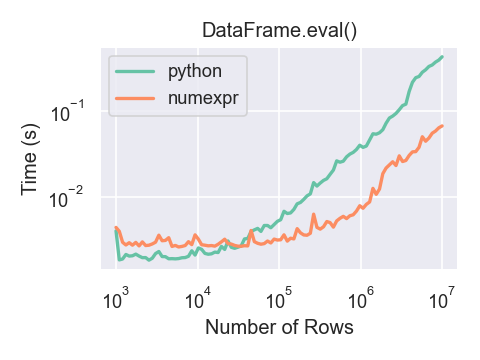
DataFrame がおよそ 100,000 行を超える場合にのみ、pandas.eval() で numexpr エンジンを使用するパフォーマンス上のメリットが得られます。
このプロットは、numpy.random.randn() を使用して生成された浮動小数点値を含む 3 つの列を持つ DataFrame を使用して作成されました。
numexpr を使用した式評価の制限#
オブジェクトdtypeになる式や、NaTを含む日時演算はPython空間で評価する必要がありますが、式の一部はnumexprで評価できます。例:
In [67]: df = pd.DataFrame(
....: {"strings": np.repeat(list("cba"), 3), "nums": np.repeat(range(3), 3)}
....: )
....:
In [68]: df
Out[68]:
strings nums
0 c 0
1 c 0
2 c 0
3 b 1
4 b 1
5 b 1
6 a 2
7 a 2
8 a 2
In [69]: df.query("strings == 'a' and nums == 1")
Out[69]:
Empty DataFrame
Columns: [strings, nums]
Index: []
比較の数値部分 (nums == 1) は numexpr によって評価され、比較のオブジェクト部分 ("strings == 'a') は Python によって評価されます。